决策树推导
首先看看下面这组数据集:
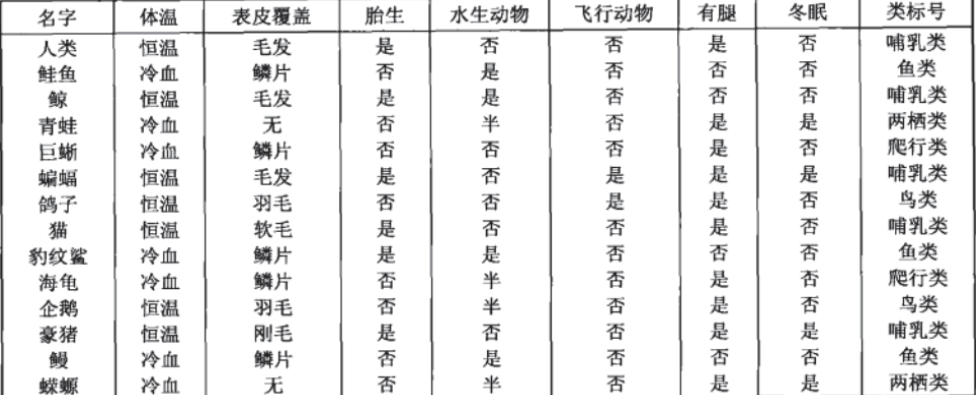
得出下面这颗决策树:

关键概念:
信息熵公式:
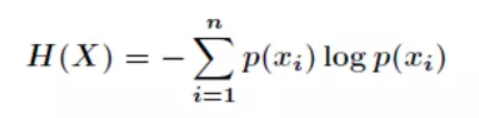
信息增益公式:就是熵和条件熵的差
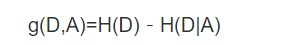
随机变量的不确定性(熵)越小,信息增益越大,这个特征的表现就越好
决策树算法的需要解决的核心问题:
1、如何从数据表中找出最佳节点和最佳分支?
2、如何让决策树停止生长,防止过拟合?
决策树的基本过程:
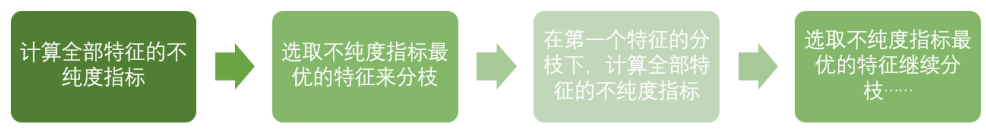
直到没有更多的特征可用,或整体的不纯度指标已经最优,决策树就会停止生长。
决策树五大模块
sklearn中决策树的类都在”tree“模块下,这个模块包含了五个类:

分类树参数、属性和接口

参数
criterion
为了要将表格转化为一棵树,决策树需要找出最佳节点和最佳的分枝方法,对分类树来说,衡量这个“最佳”的指标
叫做“不纯度”。通常来说,不纯度越低,决策树对训练集的拟合越好。现在使用的决策树算法在分枝方法上的核心
大多是围绕在对某个不纯度相关指标的最优化上。不纯度基于节点来计算,树中的每个节点都会有一个不纯度,并且子节点的不纯度一定是低于父节点的,也就是说,在同一棵决策树上,叶子节点的不纯度一定是最低的。
criterion这个参数正是用来决定不纯度的计算方法的:
1、输入”entropy“,使用信息熵
2、输入”gini“,使用基尼系数
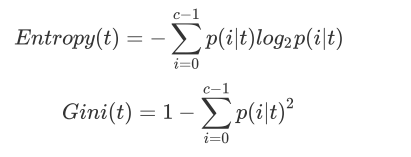
其中t代表给定的节点,i代表标签的任意分类,$p(i|t)$代表标签分类i在节点t上所占的比例。注意,当使用信息熵
时,sklearn实际计算的是基于信息熵的信息增益(Information Gain),即父节点的信息熵和子节点的信息熵之差。信息熵对不纯度更加敏感。
random_state&splitter
random_state用来设置分枝中的随机模式的参数,默认None,在高维度时随机性会表现更明显,低维度的数据
(比如鸢尾花数据集),随机性几乎不会显现。输入任意整数,会一直长出同一棵树,让模型稳定下来。
splitter也是用来控制决策树中的随机选项的,有两种输入值,输入”best”,决策树在分枝时虽然随机,但是还是会
优先选择更重要的特征进行分枝(重要性可以通过属性feature_importances_查看),输入“random”,决策树在
分枝时会更加随机,树会因为含有更多的不必要信息而更深更大,并因这些不必要信息而降低对训练集的拟合。这
也是防止过拟合的一种方式。当你预测到你的模型会过拟合,用这两个参数来帮助你降低树建成之后过拟合的可能
性。当然,树一旦建成,我们依然是使用剪枝参数来防止过拟合
max_depth
限制树的最大深度,超过设定深度的树枝全部剪掉
这是用得最广泛的剪枝参数,在高维度低样本量时非常有效。决策树多生长一层,对样本量的需求会增加一倍,所
以限制树深度能够有效地限制过拟合。在集成算法中也非常实用。实际使用时,建议从=3开始尝试,看看拟合的效
果再决定是否增加设定深度。
min_samples_leaf&min_samples_split
min_samples_leaf限定,一个节点在分枝后的每个子节点都必须包含至少min_samples_leaf个训练样本,否则分枝就不会发生,或者,分枝会朝着满足每个子节点都包含min_samples_leaf个样本的方向去发生。一般搭配max_depth使用,在回归树中有神奇的效果,可以让模型变得更加平滑。这个参数的数量设置得太小会引起过拟合,设置得太大就会阻止模型学习数据。一般来说,建议从=5开始使用。如果叶节点中含有的样本量变化很大,建议输入浮点数作为样本量的百分比来使用。同时,这个参数可以保证每个叶子的最小尺寸,可以在回归问题中避免低方差,过拟合的叶子节点出现。对于类别不多的分类问题,=1通常就是最佳选择。
min_samples_split限定,一个节点必须要包含至少min_samples_split个训练样本,这个节点才允许被分枝,否则
分枝就不会发生。
max_features&min_impurity_decrease
max_features限制分枝时考虑的特征个数,超过限制个数的特征都会被舍弃。和max_depth异曲同工,max_features是用来限制高维度数据的过拟合的剪枝参数,但其方法比较暴力,是直接限制可以使用的特征数量而强行使决策树停下的参数,在不知道决策树中的各个特征的重要性的情况下,强行设定这个参数可能会导致模型学习不足。如果希望通过降维的方式防止过拟合,建议使用PCA,ICA或者特征选择模块中的降维算法。
min_impurity_decrease限制信息增益的大小,信息增益小于设定数值的分枝不会发生。这是在0.19版本种更新的
功能,在0.19版本之前时使用min_impurity_split。
class_weight&min_weight_fraction_leaf
完成样本标签平衡的参数。样本不平衡是指在一组数据集中,标签的一类天生占有很大的比例。比如说,在银行要判断“一个办了信用卡的人是否会违约”,就是是vs否(1%:99%)的比例。这种分类状况下,即便模型什么也不做,全把结果预测成“否”,正确率也能有99%。因此我们要使用class_weight参数对样本标签进行一定的均衡,给少量的标签更多的权重,让模型更偏向少数类,向捕获少数类的方向建模。该参数默认None,此模式表示自动给与数据集中的所有标签相同的权重。有了权重之后,样本量就不再是单纯地记录数目,而是受输入的权重影响了,因此这时候剪枝,就需要搭配min_weight_fraction_leaf这个基于权重的剪枝参数来使用。另请注意,基于权重的剪枝参数(例如min_weight_fraction_leaf)将比不知道样本权重的标准(比如min_samples_leaf)更少偏向主导类。如果样本是加权的,则使用基于权重的预修剪标准来更容易优化树结构,这确保叶节点至少包含样本权重的总和的一小部分。
属性
分类树的七个参数,一个属性,四个接口,以及绘图所用的代码。
七个参数:Criterion,两个随机性相关的参数(random_state,splitter),四个剪枝参数(max_depth, min_sample_leaf,max_feature,min_impurity_decrease)
一个属性:feature_importances_ ,属性是在模型训练之后,能够调用查看的模型的各种性质。
接口
四个接口:fit,score,apply,predicd
apply:返回每个测试样本所在叶子节点的索引
predict:返回每个测试样本的分类/回归结果
coding:

回归树参数解读

几乎所有参数,属性及接口都和分类树一模一样。需要注意的是,在回归树种,没有标签分布是否均衡的问题,因此没有class_weight这样的参数。
criterion:
回归树衡量分枝质量的指标,支持的标准有三种:
1)输入”mse”使用均方误差mean squared error(MSE),父节点和叶子节点之间的均方误差的差额将被用来作为特征选择的标准,这种方法通过使用叶子节点的均值来最小化L2损失。
2)输入“friedman_mse”使用费尔德曼均方误差,这种指标使用弗里德曼针对潜在分枝中的问题改进后的均方误差
3)输入”mae”使用绝对平均误差MAE(mean absolute error),这种指标使用叶节点的中值来最小化L1损失,属性中最重要的依然是feature_importances_,接口依然是apply, fit, predict, score最核心

其中N是样本数量,i是每一个数据样本,fi是模型回归出的数值,yi是样本点i实际的数值标签。所以MSE的本质,其实是样本真实数据与回归结果的差异。在回归树中,MSE不只是我们的分枝质量衡量指标,也是我们最常用的衡量回归树回归质量的指标,当我们在使用交叉验证,或者其他方式获取回归树的结果时,我们往往选择均方误差作为我们的评估(在分类树中这个指标是score代表的预测准确率)。在回归中,我们追求的是,MSE越小越好。然而,回归树的接口score返回的是R平方,并不是MSE。R平方被定义如下 :

其中u是残差平方和(MSE * N),v是总平方和,N是样本数量,i是每一个数据样本,fi是模型回归出的数值,yi是样本点i实际的数值标签。y帽是真实数值标签的平均数。R平方可以为正为负(如果模型的残差平方和远远大于模型的总平方和,模型非常糟糕,R平方就会为负),而均方误差永远为正。值得一提的是,虽然均方误差永远为正,但是sklearn当中使用均方误差作为评判标准时,却是计算”负均方误差“(neg_mean_squared_error)。这是因为sklearn在计算模型评估指标的时候,会考虑指标本身的性质,均方误差本身是一种误差,所以被sklearn划分为模型的一种损失(loss),因此在sklearn当中,都以负数表示。真正的均方误差MSE的数值,其实就是neg_mean_squared_error去掉负号的数字。
决策树的本地保存:Graphviz
windows版本下载地址:https://graphviz.gitlab.io/_pages/Download/Download_windows.html
双击msi文件,一直next就完事了。
找到bin文件夹
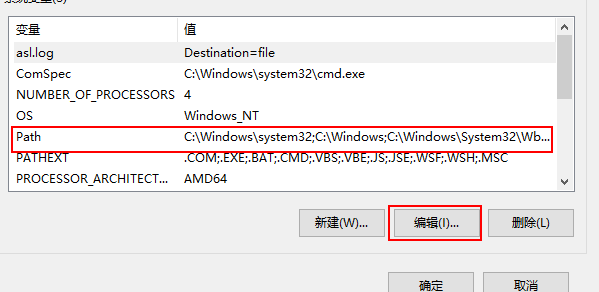
在下面这张图片的位置加入环境变量
用dot -version检查是否安装成功
将dot文件转为png文件的命令:dot -Tpng .dot -o .png

1 | #分类树 |
1 | #回归树 |

